遡河回遊の遺伝学
 近縁種群の全ゲノム配列比較により遡河回遊の起源にせまる
近縁種群の全ゲノム配列比較により遡河回遊の起源にせまる
一部の水棲生物がおこなう海と川の定期的移動、すなわち、通し回遊は、自然界に認められるもっともダイナミックな生命現象のひとつでしょう。その起源は、祖先的な海産生物が新規環境をもとめて川へ進出すること、あるいは河川の生物が海へ進出することに端を発すると考えられています。河あるいは海への進出にあたって、生物は劇的な環境の変化に直面したはずですが、この変化に耐る新しい遺伝的プログラムの獲得に成功したのが回遊生物です。回遊現象に関する研究の歴史は古く、多くの生理・行動・生態学的な知見が集積しつつあります。しかし、新規環境への進出を可能とし、回遊の発端となったゲノム上の変異は同定されていません。この興味深い問いが未解明なままである理由は、遺伝学的解析に適した回遊生物が少なかったからです。そもそも回遊するような生物は飼育するのも難しい種が多く、遺伝解析に向いていないのです。魅力的な生命現象を示す野外生物ほど研究自体が困難であるというのは、自然愛好家たちが長年抱えるジレンマでした。
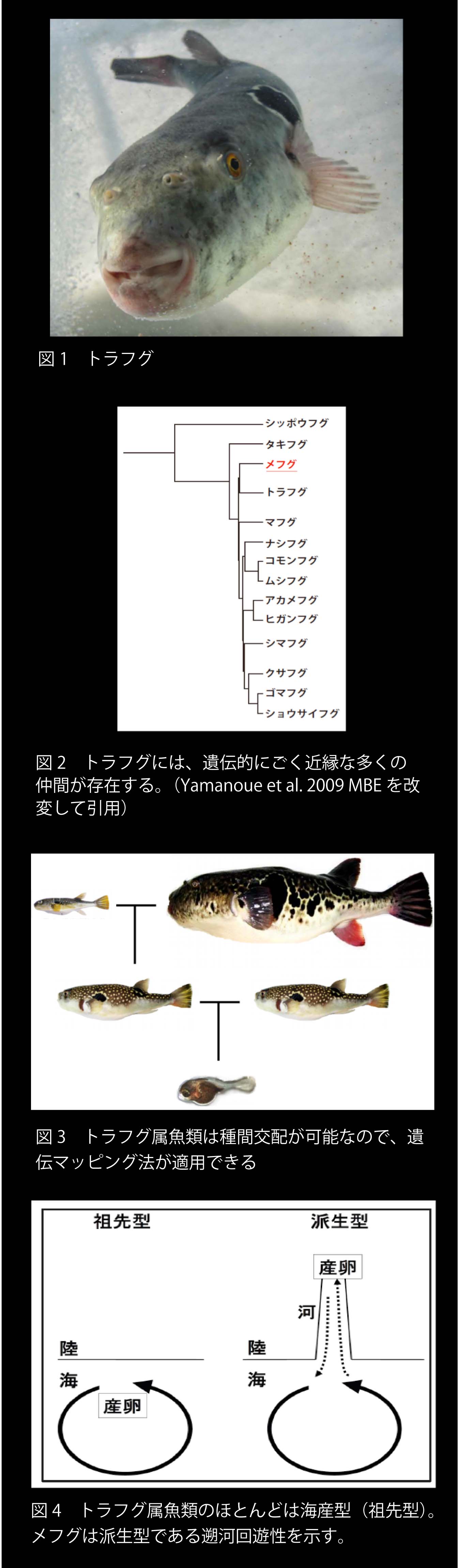
ところが、ゲノム解析技術の驚異的な進展により、状況はかわりつつあります。本研究ではトラフグとその近縁種たちの全ゲノム配列を用いた解析によって、遡河回遊を可能とした変異の同定を目指します。トラフグは我が国における代表的な海面養殖魚のひとつですが(生産額で第三位)、全ゲノム解析が進んでいるという特徴を持つ海産魚です(図1)。このトラフグには交雑が可能な約20の近縁種が存在します(図2)。そのほとんどは海でしか生きられませんが、メフグというフグは淡水への進出に成功して河でも海でも生息が可能です。「淡水に適応できる力」や「河に遡って卵を産もうとする性質」は、メフグのゲノムに変異が生じたことから生まれたはずです。この変異を、近縁種群の全ゲノム配列を比較することにより同定します。ただし、工夫もなく比較しただけでは同定できるはずはありません。「ゲノム配列を決めれば何でも分かる」といった期待もわからないではないのですが、それは大きな間違いです。種間や個体間には莫大な量のDNA配列の違いが存在しており、そのどれが表現型と結びつくかゲノム配列だけからは不明だからです。DNA配列の違いをリストアップすることは、研究の準備段階にすぎません。そして、遺伝子型と表現型を結びつける次の一手が打てるかどうかが大きなポイントとなります。次の一手が用意されていない場合、ゲノム配列情報の価値は著しく下がってしまうでしょう。幸いなことに、フグでは次の一手も二手も打ちほうだいです(言い過ぎか・・)。我々は、種間交雑を利用した遺伝マッピング(図3)、種間と種内の多様性解析といった様々な「遺伝学的」アプローチを総動員して、遡河回遊の起源にせまります。
本研究の特徴のひとつは、「陸封の起源」ではなく「遡河回遊の起源」を研究できるということにあります(図4)。例えば、モデル生物のイトヨは遡河回遊を含む様々な適応現象を研究する上で大変魅力的な生物ですが、現存する生態型は、「遡河回遊をする祖先型」と「陸封された派生型」しかないので、「陸封の起源」は問えても、「遡河回遊の起源」を問うことはできません(たぶん)(論文でMarine typeとよばれているイトヨは、実は遡河回遊型ですよね)。「回遊の起源」を問える生物種はそうはいないはずです。
方法論的な観点からみると、本研究の意義は回遊研究という分野に限定されるものではありません。将来の水産遺伝育種研究において常法となる可能性を持つアプローチ、すなわち「複数個体の全ゲノム配列比較による有用変異の同定」が本研究で確立されることが期待されます。遠くない将来に、同じ手順を踏むことにより、様々な表現型進化の原因遺伝子が多くの魚類で想定されることになるだろうと予想しています。

